PCA 視覺化高維度資料
PCA 視覺化高維度資料:
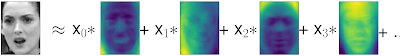
Principal components analysis 簡稱 PCA 是一種是一種分析、簡化數據集的技術。在降低數據維度的過程中我們希望能盡量保存貢獻最大的成份,經由這些貢獻最大的成份疊加我們也可以得到具有原數據特徵的數據。下圖是一個 PCA 的應用。Principal components (下圖綠色的輪廓)可以類比傅立葉的頻率,X0, X1, X2 類比傅立葉的系數。透過疊加綠色的輪廓我們可以得到某比資料(左邊灰色的圖)。
我們最大的問題其實是如何定義 Principal components,假使 Principal components 能被定義係數就只是小菜一碟。 假設我們手上有 100 張圖好了,我們想用最少的資料去紀錄這 100 張圖,我們要先找出大家共有的特徵(綠色輪廓)並對這些特徵的重要性排序,這些特徵就是 Principal components。 然後依我們的需求刪去重要性低的特徵,利用重要性高的特徵去表達整組資料。 所以 Principal components 跟傅立葉不一樣它會隨著你整組 data 的不同而有所改變,同張圖在不同資料組中會被以不同的方式表示。
下圖是一個 PCA 的原理。先來看左上的圖,考慮一組資料具有 feature1 和 feature2,我們紀錄所有 feature1 和 feature2 形成一組 dataset。如果你覺得 feature1 和 feature2 很抽象,你可以將 feature1 和 feature2 類比成鳶尾花 的花瓣長度和花瓣寬度。這組資料有兩個方向的變異度比較明顯分別是 component1 和 component2,如果我們想忽略資料的某種特徵我們會考慮拿掉 component2。
拿掉 component2 的方法其實很簡單就是旋轉,壓縮再轉回來(這句我真的不知道我在說啥XDDD)。讓我們比較一下左上和右下兩張圖,所謂的壓縮其實就是盡量維持數據原本的特徵(即變異比較大的成份)。
PCA 視覺化高維度資料範例:
PCA 視覺化高維度資料的好處是讓我們比較好去理解高維度資料糾結的狀態。我們 PCA 會選用變異度一二高的成份當成座標,如果變異度一二高的成份顯示資料沒有糾結在一起就代表資料好被分類。
#!/usr/bin/python3
# 取用 cancer data,data 經過 scaling
# 建議大家也畫畫看不經過 scaling 的資料,結果滿出人意料的
from sklearn.datasets import load_breast_cancer
from sklearn.preprocessing import StandardScaler
cancer = load_breast_cancer()
scaler = StandardScaler()
scaler.fit(cancer.data)
X_scaled = scaler.transform(cancer.data)
# PCA 分析
# 569 組 data,每個 data 有 30 個特徵
# 569 組 data,每個 data 壓縮成 2 個特徵
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca.fit(X_scaled)
X_pca = pca.transform(X_scaled)
print("X_scaled.shape: ", X_scaled.shape) # (569, 30)
print("X_pca.shape: ", X_pca.shape) #(569, 2)
# PCA 可視化資料
from matplotlib import pyplot as plt
plt.figure("PCA first and second components")
plt.xlabel("first component")
plt.ylabel("second component")
plt.axis("equal")
plt.scatter(X_pca[:, 0], X_pca[:, 1], c=cancer.target)
# 主成份1和主成份2
plt.scatter([0,1], [1,0], s=120, marker="*",c="r")
# PCA 主成份
print("pca.components_.shape: ", pca.components_.shape)
plt.matshow(pca.components_, cmap="viridis")
plt.yticks([0, 1], ["first component", "second component"])
plt.colorbar()
plt.xticks(range(len(cancer.feature_names)),
cancer.feature_names, rotation=60, ha='left')
plt.show()
PCA 後我們可以用兩個成份的係數去描述每個點相互的關係。
所謂的成份其實是原特徵的疊加,所以它的 shape 是(2, 30),上面綠色人臉圖也是一個特徵其維度和灰階圖相同。當你給定一點時我們會用成份的疊加對原資料做近似。
不 scale 的悲慘下場,每個特徵的權重差異甚大:
Reference
[1] Andreas C. Mueller and Sarah Guido, "Machine Learning with Python", O'Reilly Media (2016)






留言
張貼留言